简介
RefGene数据库是从UCSC数据库创建而来。RefGene指定了取自NCBI RNA参考序列集合(RefSeq)的已知人类蛋白质编码和非蛋白质编码的基因,用于注释变异基因。
hg19 RefGene 下载方式:
|
|
文件解析
以下是对RefGene文件各列内容的详细说明
START:0-based
END:1-based
文件示例
|
|
基因示意图
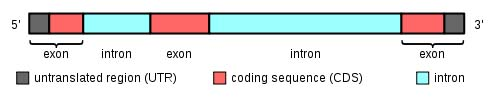
成熟mRNA示意图



